| Московский Государственный Университет
им. М. В. Ломоносова.
Факультет биоинженерии и биоинформатики
Щербинин Дмитрий Сергеевич
Компьютерная аннотация
стрептомицинового оперона бактерий
Курсовая работа
Научный руководитель:
Рассохин Т. И.
Москва
2003 г.
Аннотация
Стрептомицин - антибиотик, образующийся в процессе жизнедеятельности лучистых грибов Streptomyces globisporus streptomycini или других родственных микроорганизмов.
Стрептомицин обладает широким спектром антимикробного действия. Антибиотик активен в отношении микобактерий туберкулеза, а также большинства грамм-отрицательных бактерий (кишечная палочка, палочка Фридлендера, палочка инфлюэнцы, возбудители чумы, туляремии, бруцеллеза) и некоторых грамм-положительных (стафилококки) микроорганизмов; менее активен в отношении стрептококков, пневмококков. Не действует на анаэробы и риккетсии. Действует стрептомицин бактерицидно. Эффект связан с подавлением синтеза белка на рибосомах. Наиболее серьёзным препятствием при лечении туберкулёза - возросшая первичная (исходная) лекарственная устойчивость возбудителей заболевания: в 3 и более раза по сравнению с 60-ми годами; инфицирование населения микобактериями, приобретшими резистентность в процессе лечения. [1]
Введение
 В структуру стрептомицинового оперона E. coli
входят гены рибосомных белков малой субчастицы S12 (rpsL) и S7 (rpsG), транляционных факторов EF-G и EF-Tu. Белок S7 является регуляторным белком стрептомицинового оперона. При отсутствии свободной рРНК в клетках E.coli
белок S7 связывается с межцистронным участком S12-S7 и ингибирует синтез белков стрептомицинового оперона [2-4] (Рис. 1). Длина межцистронного фрагмента между стоп-кодоном белка S12 и старт-кодоном белка S7 – около 100 нуклеотидов. В структуру стрептомицинового оперона E. coli
входят гены рибосомных белков малой субчастицы S12 (rpsL) и S7 (rpsG), транляционных факторов EF-G и EF-Tu. Белок S7 является регуляторным белком стрептомицинового оперона. При отсутствии свободной рРНК в клетках E.coli
белок S7 связывается с межцистронным участком S12-S7 и ингибирует синтез белков стрептомицинового оперона [2-4] (Рис. 1). Длина межцистронного фрагмента между стоп-кодоном белка S12 и старт-кодоном белка S7 – около 100 нуклеотидов.
Рис. 1.
Модель вторичной структуры межцистронного фрагмента S12-S7 E
.
coli
.
Фенотипическая устойчивость E. coli
к антибиотику стрептомицину обеспечивается мутациями в гене белка S12. Поскольку стрептомицин является важным терапевтическим агентом в медицине и ветеринарии, изучение регуляции синтеза белков стрептомицинового оперона и филогенетический анализ известных бактериальных оперонов является актуальной проблемой. При общей высокой степени гомологии между рибосомными белками в различных организмах размеры и нуклеотидный состав межцистронной области, ответственной в E. coli
за регуляцию экспрессии генов оперона отличаются. Выявление общих закономерностей и установление основных регуляторных районов в стрептомициновых оперонах необходимо для полного понимания процесса регуляции и разработки новых терапевтических агентов.
Реклама

Цель данной работы – определить, является ли такой механизм аутогенной регуляции белком S7 собственного синтеза уникальным для E
.
coli
, или же существуют бактерии со схожим механизмом.
Материалы и методы
1. Построение выравнивания всех известных рибосомных белков S7 эубактерий, определение позиции старт-кодона.
В системе SRS (http://srs.ebi.ac.uk) найдены нуклеотидные последовательности изученных эубактерий*:
Actinobacteria
; Aquificiales; Cyanobacteria; Cytophagales; Spirochagales; Chlamydiales;
Thermotogales; Thermus (Deinococculus); CFB(Green sulfur); Green non-sulfur;
Pasteurellaceae; Salmonella
; Pseudomonas
; Emterobacteriaceae
; Rhodospirillaceae; Sphingomonadaceae; Rhodobacter
; Rhizobiaceae
; Ricketsiales
; Caulobacter; Bordetella;
Neisseria
; Burkholderia; Bacillus
; Clostridium
; Staphylococcus
; Heliobacterium; Mollicutes
; Streptococcus
; Enterococcus
.
Жирным шрифтом выделены найденные и использованные в работе геномы бактерий.
a) Для поиска был выбран банк EMBL.
b) В окне “Organism name” были поочерёдно введены названия соответствующих групп эубактерий (см. список бактерий)
с) В окне “Features: Gene” было введено rpsG,
d) В другом, таком же окне было введено rpsL (для того, чтобы в найденных последовательностях был не только ген белка S7, но и S12)
Записаны позиции (начало и конец) гена rpsG, учитывая, что среди найденных последовательностей могут быть как прямые (rpsL расположен до rpsG), так и комплементарные (rpsL расположен после rpsG). Для построения комплементарных последовательностей использована программа Revseq из пакета EMBOSS.
2. Экстракция нуклеотидных последовательностей области слева от старт-кодона белка S7, построение выравнивания полученных межцистронных фрагментов.
При помощи программы CutseQ из найденных последовательностей был выделен участок, отстоящий от начала rpsG на 200 нуклеотидов (т.к. близкие к str
оперону E
. с
oli
участки могут находиться как между генами S7 и S12 белков, так и в конце гена rpsL).
Используя программу Alibee
(http://www.genebee.msu.su/serv
ices/malign_reduced.html), построено выравнивания полученных участков (межцистронный фрагмент + часть rpsL)
Реклама

3. Систематизация, сортировка и аннотация найденных межцистронных фрагментов.
С помощью программы GeneDoc были построены выравнивания межцистронных областей семейств бактерий в сравнении с межцистронным фрагментом E
. coli
.
4. Построение филогенетического дерева эубактерий.
Исходя из полученных выравниваний белков S7 и межцистронных областей бактерий построены филогенетические деревья эубактерий. Для выполнения данной задачи была использована программа, расположенная на сайте http://www.genebee.msu.su/services/phtree_reduced.html
Результаты и обсуждение
В системе SRS (http://srs.ebi.ac.uk) найдены все известные геномы эубактерий. Найденные последовательности были систематизированы и унифицированы по ориентации.
При помощи программы Alibee (http://www.genebee.msu.su/services/malign_reduced.html) было построено общее выравнивание всех найденных последовательностей генов белка S7, а также участков между генами S12 и S7. При помощи этой же программы было построено филогенетическое дерево бактерий (Рис. 2, 3).
При помощи программы GeneDoc были проанализированы полученные выравнивания нуклеотидных последовательностей участков S12 - S7 белков бактерий, относящихся к одной филогенетической группе и последовательность такого же участка генома E. coli.
Выравнивание позволяет найти у изучаемых последовательностей участки, гомологичные фрагменту стрептомицинового оперона E. coli.
Рис. 2.
Филогенетическое дерево эубактерий, построенное по белку S7
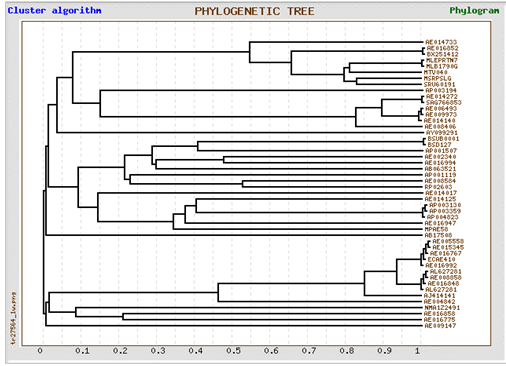
Рис. 3.
Филогенетическое дерево эубактерий, построенное по межцистронному фрагменту S12-S7.
В филогенетическом дереве, построенном по белку S7, бактерии располагаются в соответствии с их родственными связями. По построенному же по межцистронным участкам видно, что бактерии можно зразделились на 2 группы:
1) Представители, имеющие соответствующие участки, близкие к E
. с
oli
.
2) Бактерии, проявляющие значительную вариабельность данных межцистронных участков.
Найденные и использованные в работе геномы бактерии
| MLB1790G-
Mycobacterium leprae
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Corynebacterineae; Mycobacteriaceae; Mycobacterium.
|
MLEPRTN7-
Mycobacterium leprae
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Corynebacterineae; Mycobacteriaceae; Mycobacterium.
|
| MTV040-
Mycobacterium tuberculosis H37Rv
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Corynebacterineae; Mycobacteriaceae; Mycobacterium;
Mycobacterium tuberculosis complex; Mycobacterium tuberculosis.
|
MSRPSLG-
Mycobacterium smegmatis
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Corynebacterineae; Mycobacteriaceae; Mycobacterium.
|
| SRU60191-
Streptomyces roseosporus
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Streptomycineae; Streptomycetaceae; Streptomyces.
|
AE014733-
Bifidobacterium longum NCC2705
Bacteria; Actinobacteria; Actinobacteridae; Bifidobacteriales;
Bifidobacteriaceae; Bifidobacterium; Bifidobacterium longum.
|
| AE009147-
Agrobacterium tumefaciens str. C58 (U. Washington)
Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae;
Rhizobium; Agrobacterium tumefaciens; Agrobacterium tumefaciens str. C58.
|
AY099291-
Rhodobacter capsulatus
Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales;
Rhodobacteraceae; Rhodobacter.
|
| AE014017-
Buchnera aphidicola str. Bp (Baizongia pistaciae)
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Buchnera; Buchnera aphidicola;
Buchnera aphidicola (Baizongia pistaciae).
|
AP001119-
Buchnera aphidicola str. APS (Acyrthosiphon pisum)
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Buchnera; Buchnera aphidicola;
Buchnera aphidicola (Acyrthosiphon pisum).
|
| AE014125-
Buchnera aphidicola str. Sg (Schizaphis graminum)
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Buchnera; Buchnera aphidicola;
Buchnera aphidicola (Schizaphis graminum).
|
AE005558-
Escherichia coli O157:H7 EDL933
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Escherichia; Escherichia coli.
|
| AE015345-
Shigella flexneri 2a str. 301
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Shigella; Shigella flexneri; Shigella flexneri 2a.
|
AE016767-
Escherichia coli CFT073
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Escherichia; Escherichia coli.
|
| AE008858-
Salmonella typhimurium LT2
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Salmonella; Salmonella typhimurium.
|
AE016848-
Salmonella enterica subsp. enterica serovar Typhi Ty2
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Salmonella; Salmonella enterica;
Salmonella enterica subsp. enterica serovar Typhi.
|
| AL627281-
Salmonella enterica subsp. enterica serovar Typhi
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Salmonella; Salmonella enterica.
|
ECAE410-
Escherichia coli K12
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Escherichia; Escherichia coli.
|
| NMA1Z2491-
Neisseria meningitidis Z2491
Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae;
Neisseria; Neisseria meningitidis; Neisseria meningitidis serogroup A.
|
AE004842-
Pseudomonas aeruginosa PAO1
Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales;
Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa.
|
| AE016858-
Pseudomonas syringae pv. tomato str. DC3000
Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales;
Pseudomonadaceae; Pseudomonas; Pseudomonas syringae;
Pseudomonas syringae pv. tomato.
|
AE016775-
Pseudomonas putida KT2440
Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales;
Pseudomonadaceae; Pseudomonas; Pseudomonas putida.
|
| AE016852-
Tropheryma whipplei str. Twist
Bacteria; Actinobacteria; Tropheryma; Tropheryma whipplei.
|
BX251412-
Tropheryma whipplei TW08/27
Bacteria; Actinobacteria; Tropheryma; Tropheryma whipplei.
|
| AP001507-
Bacillus halodurans
Bacteria; Firmicutes; Bacillales; Bacillaceae; Bacillus.
|
BSD127-
Bacillus subtilis
Bacteria; Firmicutes; Bacillales; Bacillaceae; Bacillus.
|
| AE016947-
Enterococcus faecalis V583
Bacteria; Firmicutes; Lactobacillales; Enterococcaceae; Enterococcus;
Enterococcus faecalis.
|
AP003130-
Staphylococcus aureus subsp. aureus N315
Bacteria; Firmicutes; Bacillales; Staphylococcus; Staphylococcus aureus;
Staphylococcus aureus subsp. aureus.
|
| AP003359-
Staphylococcus aureus subsp. aureus Mu50
Bacteria; Firmicutes; Bacillales; Staphylococcus; Staphylococcus aureus;
Staphylococcus aureus subsp. aureus.
|
AP004823-
Staphylococcus aureus subsp. aureus MW2
Bacteria; Firmicutes; Bacillales; Staphylococcus; Staphylococcus aureus;
Staphylococcus aureus subsp. aureus.
|
| AE006493-
Streptococcus pyogenes M1 GAS
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus pyogenes.
|
AE009973-
Streptococcus pyogenes MGAS8232
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus pyogenes.
|
| AE014140-
Streptococcus pyogenes MGAS315
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus pyogenes.
|
AE014272-
Streptococcus agalactiae 2603V/R
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus agalactiae; Streptococcus agalactiae serogroup V.
|
| SAG766853-
Streptococcus agalactiae NEM316
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus agalactiae; Streptococcus agalactiae serogroup III.
|
AE008406-
Streptococcus pneumoniae R6
Bacteria; Firmicutes; Lactobacillales; Streptococcaceae; Streptococcus;
Streptococcus pneumoniae.
|
| AP003194-
Clostridium perfringens str. 13
Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae;
Clostridium; Clostridium perfringens.
|
MPAE58-
Mycoplasma pneumoniae
Bacteria; Firmicutes; Mollicutes; Mycoplasmataceae; Mycoplasma.
|
| AE002340-
Chlamydia muridarum
Bacteria; Chlamydiae; Chlamydiales; Chlamydiaceae; Chlamydia.
|
AE016994-
Chlamydophila caviae GPIC
Bacteria; Chlamydiae; Chlamydiales; Chlamydiaceae; Chlamydophila;
Chlamydophila caviae.
|
| AE008584-
Rickettsia conorii
Bacteria; Proteobacteria; Alphaproteobacteria; Rickettsiales;
Rickettsiaceae; Rickettsieae; Rickettsia; spotted fever group.
|
RP02603-
Rickettsia prowazekii
Bacteria; Proteobacteria; Alphaproteobacteria; Rickettsiales;
Rickettsiaceae; Rickettsieae; Rickettsia; typhus group.
|
| BSUB0001-
Bacillus subtilis subsp. subtilis str. 168
Bacteria; Firmicutes; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis;
Bacillus subtilis subsp. subtilis.
|
AB017508-
Bacillus halodurans
Bacteria; Firmicutes; Bacillales; Bacillaceae; Bacillus.
|
Далее приведены выравнивания фрагментов с наибольшей степенью гомологии. В участках геномов остальных групп бактерий расхождение с E
. с
oli
значительно больше.
Рис. 4
. Выравнивание межцистронных фрагментов S7-S12 из геномов, наиболее схожих с аналогичным участком генома E
.
coli
. Фрагменты заканчиваются перед старт-кодоном белка S7.
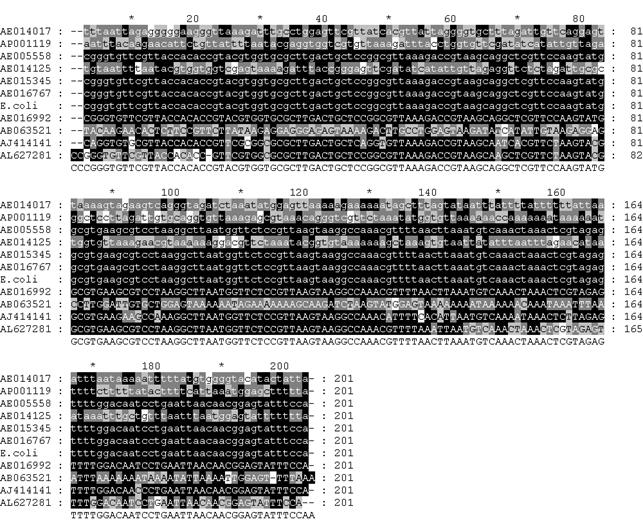
Рис. 5.
Выравнивание групп бактерий, наиболее схожих с E
.
coli
по составу межцистронных фрагментов:
a) Enterobacteriaceae
(в эту группу входит E
.
coli
)
Фрагменты заканчиваются перед старт-кодоном белка S7.

Рис. 5.
Выравнивание групп бактерий, наиболее схожих с E
.
coli
по составу межцистронных фрагментов:
б) Salmonella
Фрагменты заканчиваются перед старт-кодоном белка S7.
По данным проведенного анализа можно предположить, что у бактерий семейства Enterobacteriaceae
и Salmonella
механизм аутогенной регуляции белком S7 собственного синтеза подобен E
.
coli
, т.е. межцистронный фрагмент S12-S7 связывается с белком S7. Высокая степень гомологии соответствующих участков геномов бактерий группы Enterobacteriaceae
связана только с генетической близостью данных представителей, и не доказывает, что механизм соответствующей регуляции других организмов идентичен E
. с
oli
.
Например, почти полное отсутствие межцистронного участка у бактерий группы Bacillus
свидетельствует о возможности другого механизма регуляции. Косвенное подтверждение этой возможности было получены биохимическими методами [1]
Выводы
1. Не смотря на высокую степень гомологии белков S7 эубактерий, межцистронные фрагменты S12-S7 str
оперона демонстрируют значительную вариабельность.
2. Наиболее близкие к str
оперону E
. с
oli
участки имеются у членов семейства Enterobacteriaceae
, к которым относится E. coli
, и у Salmonella
. Можно предположить, что механизм аутогенной регуляции белком S7 собственного синтеза у E
.
coli
также присутствует и у бактерий семейства Salmonella
.
3. Практически полное отсутствие межцистронной области S12-S7 в str
оперонах бактерий группы Bacillus
свидетельствует об отличном от E
.
coli
механизме регуляции.
Список литературы
[1] Miyamoto, A., Usui, M., Yamasaki, N., Yamada, N., Kuwano, E., Tanaka, I. and Kimura, M. (1999) Eur J Biochem 266, 591-8.
[2] Saito, K., Mattheakis, L.C. and Nomura, M. (1994) J Mol Biol 235, 111-24.
[3] Saito, K. and Nomura, M. (1994) J Mol Biol 235, 125-39.
[4] Zengel, J.M. and Lindahl, L. (1994) Prog Nucleic Acid Res Mol Biol 47, 331-70.
|